Comentarii Adauga Comentariu
_ O nouă tehnică de calcul ar putea facilita proiectarea proteinelor utile
_ O nouă tehnică de calcul ar putea face mai ușoară proiectarea proteinelor utile
Pentru a proiecta proteine cu funcții utile, cercetătorii încep de obicei cu o proteină naturală care are o funcție dorită, cum ar fi emiterea de lumină fluorescentă, și o pun prin multe runde de mutații aleatorii care în cele din urmă generează o versiune optimizată a proteinei.
Acest proces a dat versiuni optimizate ale multor proteine importante, inclusiv proteina verde fluorescentă (GFP). Cu toate acestea, pentru alte proteine, sa dovedit dificil să se genereze o versiune optimizată. Cercetătorii MIT au dezvoltat acum o abordare computațională care facilitează prezicerea mutațiilor care vor duce la proteine mai bune, pe baza unei cantități relativ mici de date.
Folosind acest model, cercetătorii au generat proteine cu mutații care au fost se prevede că va duce la versiuni îmbunătățite de GFP și o proteină din virusul adeno-asociat (AAV), care este folosită pentru a furniza ADN pentru terapia genică. Ei speră că ar putea fi folosit și pentru a dezvolta instrumente suplimentare pentru cercetarea în neuroștiință și aplicații medicale.
„Designul proteinelor este o problemă grea, deoarece maparea de la secvența ADN la structura și funcția proteinelor este cu adevărat complexă. o proteină grozavă 10 se schimbă în secvență, dar fiecare modificare intermediară ar putea corespunde unei proteine total nefuncționale.
„Este ca și cum ai încerca să-ți găsești drumul către bazinul râului într-un lanț muntos, când sunt stâncoși. vârfuri de-a lungul drumului care vă blochează vederea. Lucrarea actuală încearcă să facă albia mai ușor de găsit”, spune Ila Fiete, profesor de științe ale creierului și cognitive la MIT, membru al Institutului McGovern pentru Cercetare a Creierului al MIT, director al Centrului Integrativ de Neuroscience Computațională K. Lisa Yang și unul dintre autorii principali ai studiului.
Regina Barzilay, profesor distins la Școala de Inginerie pentru AI și sănătate la MIT, și Tommi Jaakkola, profesor Thomas Siebel de inginerie electrică și informatică la MIT, sunt De asemenea, autori seniori ai unei lucrări cu acces deschis despre lucrare, care va fi prezentată la Conferința Internațională privind Reprezentațiile Învățării (ICLR 2024) în mai. Este disponibilă pe serverul arXiv preprint.
Studenții absolvenți ai MIT Andrew Kirjner și Jason Yim sunt autorii principali ai studiului. Alți autori includ Shahar Bracha, un post-doctorat MIT și Raman Samusevich, un student absolvent la Universitatea Tehnică Cehă.
Multe proteine din natură au funcții care ar putea fă-le utile pentru cercetare sau aplicații medicale, dar au nevoie de puțină inginerie suplimentară pentru a le optimiza. În acest studiu, cercetătorii au fost inițial interesați de dezvoltarea proteinelor care ar putea fi utilizate în celulele vii ca indicatori de tensiune.
Aceste proteine, produse de unele bacterii și alge, emit lumină fluorescentă atunci când este detectat un potențial electric. Dacă sunt concepute pentru a fi utilizate în celulele de mamifere, astfel de proteine ar putea permite cercetătorilor să măsoare activitatea neuronilor fără a utiliza electrozi.
În timp ce zeci de ani de cercetare s-au dus în proiectarea acestor proteine pentru a produce un semnal fluorescent mai puternic, la o scară de timp mai rapidă, nu au devenit suficient de eficiente pentru a fi utilizate pe scară largă. Bracha, care lucrează în laboratorul lui Edward Boyden de la Institutul McGovern, a contactat laboratorul lui Fiete pentru a vedea dacă ar putea lucra împreună la o abordare computațională care ar putea ajuta la accelerarea procesului de optimizare a proteinelor.
„Această lucrare exemplifică serendipitatea umană care caracterizează atâtea descoperiri științifice”, spune Fiete. „A apărut din retragerea Yang Tan Collective, o întâlnire științifică a cercetătorilor din mai multe centre de la MIT cu misiuni distincte unificate prin sprijinul comun al lui K. Lisa Yang. Am aflat că unele dintre interesele și instrumentele noastre în modelarea modului în care creierul învață și optimize ar putea fi aplicat în domeniul total diferit al designului proteinelor, așa cum este practicat în laboratorul Boyden.”
Pentru orice proteină dată pe care cercetătorii ar putea dori să o optimizeze, există un număr aproape infinit de secvențe posibile care ar putea fi generată prin schimbarea diferiților aminoacizi în fiecare punct din secvență. Cu atât de multe variante posibile, este imposibil să le testăm pe toate experimental, așa că cercetătorii au apelat la modelarea computațională pentru a încerca să prezică care dintre ele vor funcționa cel mai bine.
În acest studiu, cercetătorii și-au propus să depășească acele provocări, folosind date de la GFP pentru a dezvolta și testa un model de calcul care ar putea prezice versiuni mai bune ale proteinei.
Ei au început prin a antrena un tip de model cunoscut sub numele de rețea neuronală convoluțională (CNN) pe date experimentale. constând din secvențe GFP și luminozitatea acestora – caracteristica pe care doreau să o optimizeze.
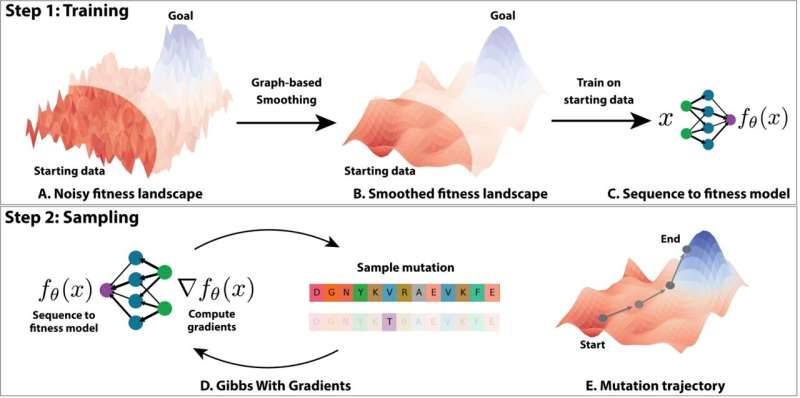
Modelul a reușit să creeze un „peisaj de fitness” – o hartă tridimensională care descrie fitness-ul unei anumite proteine și modul în care diferă mult de secvența originală – bazată pe o cantitate relativ mică de date experimentale (de la aproximativ 1.000 de variante de GFP).
Aceste peisaje conțin vârfuri care reprezintă proteine mai potrivite și văi care reprezintă proteine mai puțin potrivite. Prezicerea căii pe care trebuie să o urmeze o proteină pentru a atinge vârfurile de fitness poate fi dificilă, deoarece adesea o proteină va trebui să sufere o mutație care o face mai puțin potrivită înainte de a atinge un vârf apropiat de fitness mai înalt. Pentru a depăși această problemă, cercetătorii au folosit o tehnică de calcul existentă pentru a „netezi” peisajul fitnessului.
Odată ce aceste mici denivelări din peisaj au fost netezite, cercetătorii au reantrenat modelul CNN și au descoperit că acesta a fost capabil să depășească această problemă. atinge mai ușor vârfuri de fitness mai mari. Modelul a fost capabil să prezică secvențe GFP optimizate care aveau până la șapte aminoacizi diferiți din secvența de proteine cu care au început, iar cele mai bune dintre aceste proteine au fost estimate a fi de aproximativ 2,5 ori mai potrivite decât originalul.
„Odată ce avem acest peisaj care reprezintă ceea ce modelul crede că este în apropiere, îl netezim și apoi reantrenăm modelul pe versiunea mai netedă a peisajului”, spune Kirjner. „Acum există o cale lină de la punctul de pornire până la vârf, pe care modelul este acum capabil să o atingă făcând iterativ mici îmbunătățiri. Același lucru este adesea imposibil pentru peisajele nenetezite.”
Cercetătorii au mai arătat. că această abordare a funcționat bine în identificarea de noi secvențe pentru capside virală a virusului adeno-asociat (AAV), un vector viral care este utilizat în mod obișnuit pentru a furniza ADN. În acest caz, au optimizat capsida pentru capacitatea sa de a împacheta o încărcătură utilă ADN.
„Am folosit GFP și AAV ca dovadă de concept pentru a arăta că aceasta este o metodă care funcționează pe seturi de date foarte bine caracterizată și, din această cauză, ar trebui să fie aplicabilă altor probleme de inginerie a proteinelor", spune Bracha.
Cercetătorii intenționează acum să utilizeze această tehnică de calcul pe datele pe care Bracha le-a generat asupra proteinelor indicator de tensiune. .
„Zezi de laboratoare au lucrat la asta timp de două decenii și încă nu există nimic mai bun”, spune ea. „Speranța este că acum, odată cu generarea unui set de date mai mic, am putea antrena un model in silico și am putea face predicții care ar putea fi mai bune decât ultimele două decenii de testare manuală.”
Această poveste este republicată prin curtoazie. de MIT News (web.mit.edu/newsoffice/), un site popular care acoperă știri despre cercetarea, inovarea și predarea MIT.























































Comentarii:
Adauga Comentariu